solo para uso en investigación
SU 5402 Inhibidor de Tirosina Quinasa
Cat. No.S7667
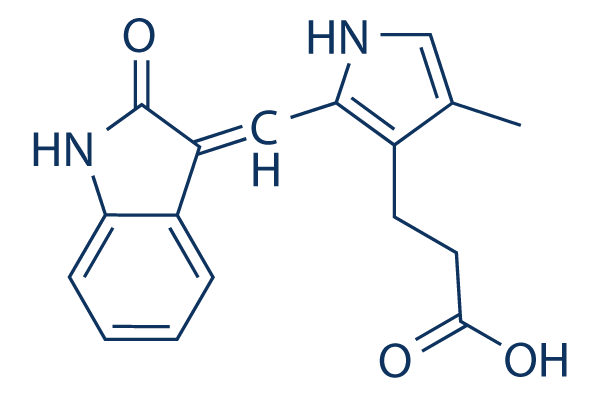
Estructura química
Peso molecular: 296.32
Control de calidad
| Dianas relacionadas | EGFR PDGFR FGFR c-Met Src MEK CSF-1R FLT3 HER2 c-Kit |
|---|---|
| Otros VEGFR Inhibidores | SAR131675 Cediranib (AZD2171) Vatalanib (PTK787) 2HCl Anlotinib (AL3818) Dihydrochloride Linifanib (ABT-869) Apatinib (YN968D1) Apatinib (YN968D1) mesylate Ki8751 ZM 323881 HCl Semaxanib (SU5416) |
Cultivo celular, tratamiento y concentración de trabajo
| Líneas celulares | Tipo de ensayo | Concentración | Tiempo de incubación | Formulación | Descripción de la actividad | PMID |
|---|---|---|---|---|---|---|
| HUVEC or NIH3T3 | Function assay | Evaluated for the inhibition of cell proliferation induced by VEGF in HUVEC or NIH3T3 cells, IC50=0.05μM | 10893303 | |||
| HUVEC or NIH3T3 | Function assay | Evaluated for the inhibition of cell proliferation induced by FGF in HUVEC or NIH3T3 cells, IC50=2.8μM | 10893303 | |||
| NIH/3T3 | Function assay | Inhibition of acidic FGF-stimulated FGFR1 tyrosine autophosphorylation in mouse NIH/3T3 cells by immunoblotting, IC50=10μM | 9139660 | |||
| NIH 3T3 | Function assay | 5 mins | Inhibition of FGF induced FGFR1 autophosphorylation in mouse NIH 3T3 cells preincubated for 5 mins followed by FGF-stimulation for 5 mins in presence of [gamma-32P]ATP by SDS-PAGE based autoradiography, IC50=10μM | 27914362 | ||
| HUVEC or NIH3T3 | Function assay | Evaluated for the inhibition of cell proliferation induced by PDGF in HUVEC or NIH3T3 cells, IC50=28.4μM | 10893303 | |||
| NIH/3T3 | Growth inhibition assay | Growth inhibition of mouse NIH/3T3 cells assessed as inhibition of acidic FGF-stimulated [3H]thymidine incorporation | 9139660 | |||
| NIH/3T3 | Function assay | Inhibition of FGFR1 in mouse NIH/3T3 cells assessed as inhibition of aFGF-stimulated pp90 phosphoprotein phosphorylation by immunoblotting | 9139660 | |||
| NIH/3T3 | Function assay | Inhibition of FGFR1 in mouse NIH/3T3 cells assessed as inhibition of acidic FGF-stimulated ERK1 phosphorylation by immunoblotting | 9139660 | |||
| NIH/3T3 | Function assay | Inhibition of FGFR1 in mouse NIH/3T3 cells assessed as inhibition of acidic FGF-stimulated ERK2 phosphorylation by immunoblotting | 9139660 | |||
| NIHIR | Function assay | Inhibition of insulin-stimulated insulin receptor beta subunit autophosphorylation in mouse NIHIR cells | 9139660 | |||
| HER14 | Function assay | Inhibition of EGF-stimulated EGFR phosphorylation in mouse HER14 cells by immunoblotting | 9139660 | |||
| HER14 | Function assay | Inhibition of EGFR in mouse HER14 cells assessed as inhibition of EGF-stimulated Shc phosphorylation by immunoblotting | 9139660 | |||
| HER14 | Function assay | Inhibition of EGFR in mouse HER14 cells assessed as inhibition of EGF-stimulated ERK2 activation by immunoblotting | 9139660 | |||
| NIHIR | Function assay | Inhibition of insulin receptor in mouse NIHIR cells assessed as inhibition of insulin-stimulated IRS1 phosphorylation | 9139660 | |||
| NIH/3T3 | Function assay | Inhibition of PDGFR in mouse NIH/3T3 cells assessed as inhibition of PDGF-stimulated tyrosine autophosphorylation by immunoblotting | 9139660 | |||
| NIH/3T3 | Function assay | Inhibition of PDGFR in mouse NIH/3T3 cells assessed as inhibition of PDGF-stimulated phospholipase C gamma phosphorylation by immunoblotting | 9139660 | |||
| NIH/3T3 | Function assay | Inhibition of PDGFR in mouse NIH/3T3 cells assessed as PDGF-stimulated Erk2 activation by immunoblotting | 9139660 | |||
| Haga clic para ver más datos experimentales de líneas celulares | ||||||
Información química, almacenamiento y estabilidad
| Peso molecular | 296.32 | Fórmula | C17H16N2O3 |
Almacenamiento (Desde la fecha de recepción) | |
|---|---|---|---|---|---|
| Nº CAS | 215543-92-3 | Descargar SDF | Almacenamiento de soluciones madre |
|
|
| Sinónimos | N/A | Smiles | CC1=CNC(=C1CCC(=O)O)C=C2C3=CC=CC=C3NC2=O | ||
Solubilidad
|
In vitro |
DMSO
: 59 mg/mL
(199.1 mM)
Water : Insoluble Ethanol : Insoluble |
Calculadora de Molaridad
|
In vivo |
|||||
Calculadora de formulación in vivo (Solución clara)
Paso 1: Introduzca la información a continuación (Recomendado: Un animal adicional para tener en cuenta la pérdida durante el experimento)
Paso 2: Introduzca la formulación in vivo (Esto es solo la calculadora, no la formulación. Por favor, contáctenos primero si no hay una formulación in vivo en la sección de Solubilidad.)
Resultados del cálculo:
Concentración de trabajo: mg/ml;
Método para preparar el líquido maestro de DMSO: mg fármaco predissuelto en μL DMSO ( Concentración del líquido maestro mg/mL, Por favor, contáctenos primero si la concentración excede la solubilidad del DMSO del lote del fármaco. )
Método para preparar la formulación in vivo: Tomar μL DMSO líquido maestro, luego añadirμL PEG300, mezclar y clarificar, luego añadirμL Tween 80, mezclar y clarificar, luego añadir μL ddH2O, mezclar y clarificar.
Método para preparar la formulación in vivo: Tomar μL DMSO líquido maestro, luego añadir μL Aceite de maíz, mezclar y clarificar.
Nota: 1. Por favor, asegúrese de que el líquido esté claro antes de añadir el siguiente disolvente.
2. Asegúrese de añadir el (los) disolvente(s) en orden. Debe asegurarse de que la solución obtenida, en la adición anterior, sea una solución clara antes de proceder a añadir el siguiente disolvente. Se pueden utilizar métodos físicos como el vórtice, el ultrasonido o el baño de agua caliente para ayudar a la disolución.
Mecanismo de acción
| Targets/IC50/Ki |
VEGFR2
(Cell-free assay) 20 nM
FGFR1
(Cell-free assay) 30 nM
PDGFRβ
(Cell-free assay) 510 nM
|
|---|---|
| In vitro |
SU5402 inhibe la proliferación celular dependiente de VEGF, FGF, PDGF con una IC50 de 0,05 μM, 2,80 μM y 28,4 μM, respectivamente. En HUVECs, este compuesto inhibe selectivamente la mitogénesis impulsada por VEGF de manera dosis-dependiente con una IC50 de 0,04 μM. En células epiteliales nasofaríngeas, atenúa la glucólisis aeróbica mediada por LMP1, la transformación celular, la migración celular y la invasión. En células de ratón C3H10T1/2, este químico disminuye el efecto de FGF23 sobre la diferenciación celular.
|
| Ensayo de quinasa |
Ensayos de quinasa de FGF-R1 y Flk-1/KDR.
|
|
La porción catalítica de FGF-R1 y Flk-1/KDR se expresa como proteínas de fusión GST después de la infección de células de Spodoptera frugiperda (sf9) con baculovirus modificados. GST-FGFR1 y GST-Flk1 se purifican hasta la homogeneidad a partir de lisados de células sf9 infectadas mediante cromatografía de sefarosa de glutatión. Los ensayos se realizan en microplacas de 96 pocillos que se habían recubierto durante la noche con 2,0 μg de un péptido poliGlu-Tyr (4:1) en 0,1 mL de PBS por pocillo. Las quinasas purificadas se diluyen en tampón de ensayo de quinasa (100 mM Hepes pH 7,5, 100 mM NaCl y 0,1 mM ortovanadato de sodio) y se añaden a todos los pocillos de prueba a 5 ng de proteína de fusión GST por 0,05 mL de volumen de tampón. Los compuestos de prueba se diluyen en 4% de DMSO y se añaden a los pocillos de prueba (0,025 mL/pocillo). La reacción de la quinasa se inicia con la adición de 0,025 mL de 40 μM ATP/40 mM MnCl2, y las placas se agitan durante 10 min antes de detener las reacciones con la adición de 0,025 mL de 0,5 M EDTA. La concentración final de ATP fue de 10 μM, que es el doble del valor de Km determinado experimentalmente para el ATP. Los pocillos de control negativo reciben solo MnCl2 sin ATP. Las placas se lavan tres veces con 10 mM Tris pH 7,4, 150 mM NaCl y 0,05% Tween-20 (TBST). Se añade antisérum policlonal de conejo anti-fosfotirosina a los pocillos en una dilución de 1:10000 en TBST durante 1 h. Las placas se lavan luego tres veces con TBST. Se añadió entonces antisérum de cabra anti-conejo conjugado con peroxidasa de rábano picante a todos los pocillos durante 1 h. Las placas se lavan tres veces con TBST, y la reacción de la peroxidasa se detecta con la adición de 2,2'-azinobis(ácido 3-etilbenzotiazolina-6-sulfónico) (ABTS). Se permite que la lectura de color del ensayo se desarrolle durante 20-30 min y se lee en un lector de placas ELISA Dynatech MR5000 usando un filtro de prueba de 410 nM.
|
|
| In vivo |
En ratones, SU5416 (25 mg/kg, i.p.) inhibe el crecimiento subcutáneo de un panel de líneas celulares tumorales al inhibir el proceso angiogénico asociado al crecimiento tumoral.
|
Referencias |
|
Soporte técnico
Tel: +1-832-582-8158 Ext:3
Si tiene alguna otra consulta, por favor deje un mensaje.
